应用数学与交叉科学研究中心生物信息学团队于2023年10月第3周组会按期举行,小组全体成员和各位导师共同参加。在这次组会上,由一名研三员工和两名研二员工分别汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
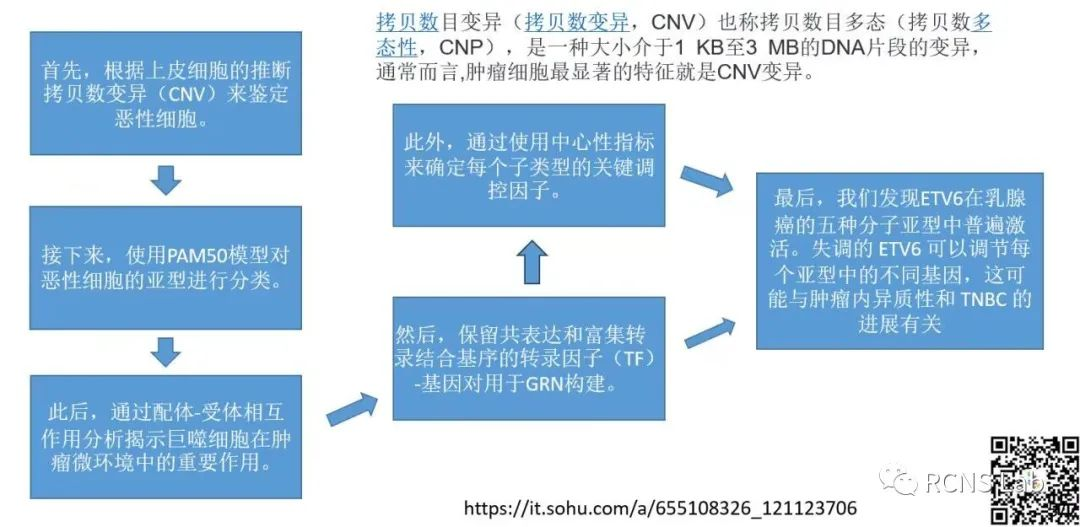
王梦园:本次汇报了一篇文献《Single-cell RNA-seq dissects the intratumoral heterogeneity of triple-negative breast cancer based on gene regulatory networks》。最近,Lacono等人应用全局调控模型构建了基于scRNA-seq数据的基因调控网络(GRN),这可以帮助我们识别疾病的关键调控因子。因此,基于scRNA-seq数据的TNBC网络分析可以帮助我们剖析在肿瘤发生中起关键作用的风险基因。在这项研究中,本文对乳腺癌亚型的 GRN 进行全面分析,并剖析了每种分子亚型的关键基因。
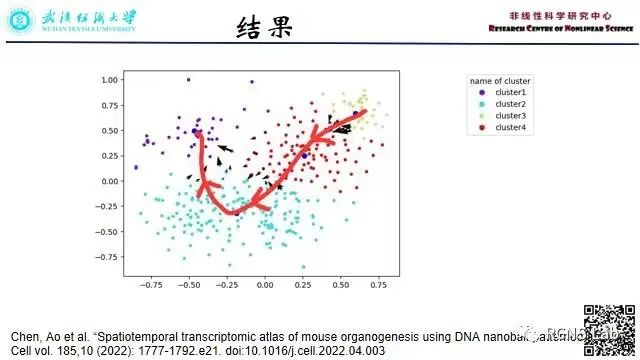
孙睿:本次汇报了自己最新的研究进展,通过构建随机游走模型和空间吸引子来描述细胞的分化行为,给细胞分化的研究提供了另外一条可行的方法;展示了模型结果的3D可视化,对现在面临的问题和老师进行了讨论。
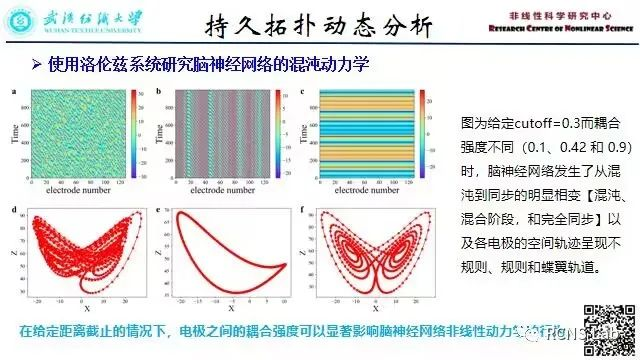
柯璐:本次组会汇报的是研究进展,主题是《研究框架与部分结果分析》。本研究提出了一个新框架,通过分析系统中单个组件的拓扑结构来预测系统的物理和化学特性。这为从混沌模型进行拓扑机器学习预测的方法论带来了新的启示,从而可以更好地描述真实世界数据(尤其是大脑和生物分子数据)固有的个体动态特性。将方法应用于脑电图EEG数据,发现了三种不同的混沌动力学模式,即混沌、混沌与同步的混合以及同步。此外,我们的方法还得到了由 364 个蛋白质组成的基准集的 B 因子预测的验证,优于现有的其他最先进的方法将平均 PCCs最多提高 49.6%。
— 员工汇报照片展示 —


