应用数学与交叉科学研究中心生物信息学团队于2023年12月19日举行第三十八期研究生论坛,小组全体成员和各位导师共同参加。在这次组会上,由一名研一员工和两名研二员工分别汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
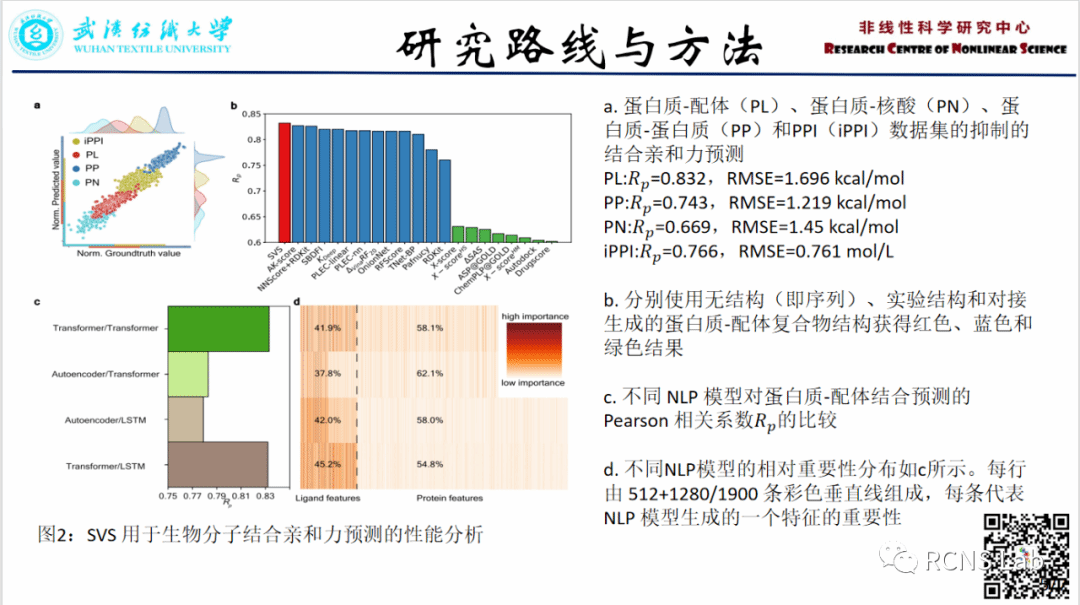
陈龙:本次汇报了一篇文献《SVSBI: sequence-based virtual screening of biomolecular interactions》。虚拟筛选(VS)是理解生物分子相互作用的一项关键技术,特别是在药物设计和发现方面。然而,当前VS模型的精度严重依赖于通过分子对接获得的三维(3D)结构,由于精度较低,通常不可靠。为了解决这个问题,我们引入了基于序列的虚拟筛选(SVS)作为另一代VS模型,它利用先进的自然语言处理(NLP)算法和优化的深度K嵌入策略来编码生物分子相互作用,而不依赖于基于3D结构的对接。我们证明,对于涉及蛋白质-配体结合、蛋白质-蛋白质、蛋白质-核酸结合和蛋白质-蛋白质相互作用的配体抑制的四个回归数据集以及蛋白质-蛋白质相互作用的五个分类数据集,SVS 的性能优于最先进的性能在五个生物物种中。SVS 有潜力改变药物发现和蛋白质工程的当前实践。
孙睿:本次汇报了一篇文献《Dimension-agnostic and granularity-based spatially variable gene identification using BSP》。识别空间可变基因(SVGs)是连接分子细胞功能与组织表型的关键。空间解析转录组学在二维或三维空间坐标上捕获细胞水平的基因表达,可以有效地推断svg。然而,目前的计算方法可能无法获得可靠的结果,并且往往无法处理三维空间转录组数据。在这里,我们引入了一种非参数模型BSP (big-small patch),通过比较两个空间粒度的基因表达模式,以快速和稳健的方式从二维或三维空间转录组学数据中识别svg。该方法已在仿真中进行了广泛的测试,证明了优越的准确性、鲁棒性和高效率。在癌症、神经科学、类风湿关节炎和肾脏研究中,利用各种类型的空间转录组学技术进一步证实了BSP的生物学发现。

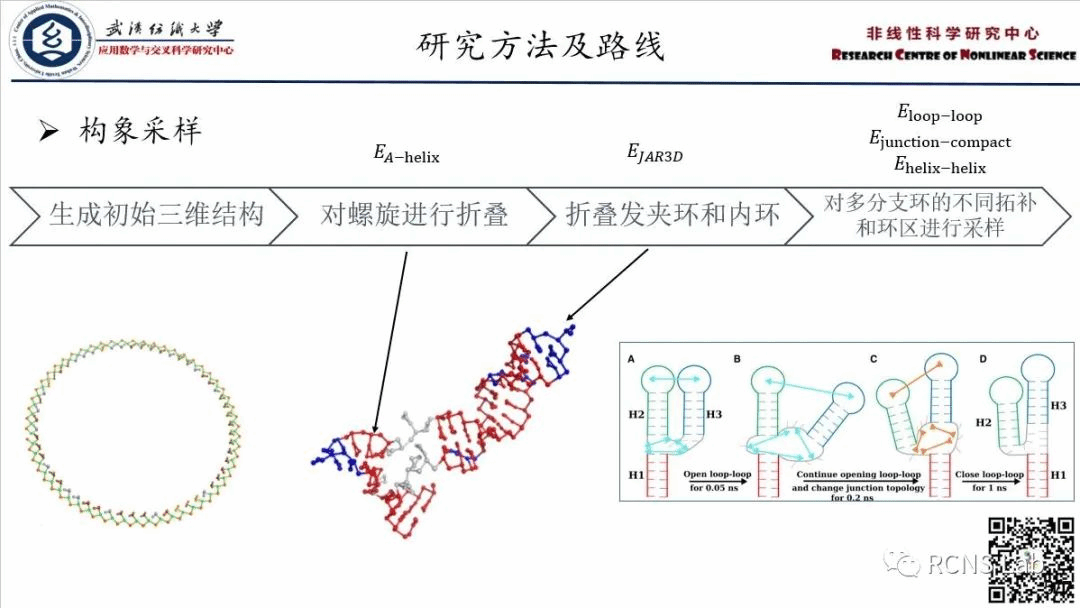
李莎莎:本次汇报了一篇文献《RNAJP: enhanced RNA 3D structure predictions with non-canonical interactions and global topology sampling》。RNA 3D结构对于理解其功能至关重要。然而,只有有限数量的RNA结构已被实验解决。由于RNA分子内复杂的各种相互作用,通过计算方法准确预测RNA的3D结构,特别是那些含有多分支环的,仍然是一个重大的挑战。因此该文献提出了一个核苷酸和螺旋水平的粗粒度模型RNAJP(“RNA Junction Prediction”),用于从给定的2D结构预测RNA的3D结构,特别是多分支环结构。通过使用分子动力学模拟和明确考虑非规范碱基配对和碱基堆叠相互作用以及长程环-环相互作用等能量约束,对多分支环进行全局拓扑采样,该模型比现有方法具有显著改进。此外,结合实验的额外约束,如多分支环拓扑结构和长程相互作用,该模型可以在各种应用中作为一个有用的结构生成器。
— 员工汇报照片展示 —


