应用数学与交叉科学研究中心生物信息学团队于2023年10月第4周组会按期举行,小组全体成员和各位导师共同参加。在这次组会上,由一名研三员工和三名研二员工分别汇报自己的研究进展,然后老师与同学们对汇报内容进行学术探讨,并对存在的问题给出相应的指导和建议。
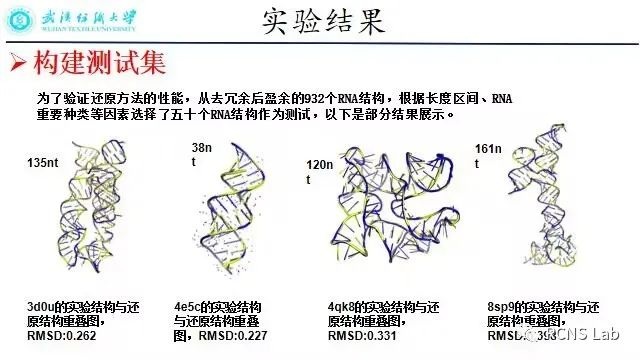
吴昊:本次汇报了自己的工作总结,CGAA是一种针对于粗粒化Rna模型的全原子还原工具,从目前已知的符合要求的RNA结构中通过计算rmsd的方式筛选出了A、U、C、G核苷酸的各种典型模板。通过SVD分解推导出典型模板核苷酸中重点原子到粗粒化核苷酸原子的旋转矩阵,从而将典型模板中其它原子同步还原至粗粒化核苷酸,从而完成单个粗粒化核苷酸的还原,继而还原整个Rna结构。与其它还原工具相比较CGAA有良好的还原性能。
陈龙:本次汇报了一篇文献《Multiobjective Molecular Optimization for Opioid Use Disorder Treatment Using Generative Network Complex》,阿片类药物使用障碍(OUD)已成为一个重大的全球公共卫生问题,因此需要发现新的药物。在这项研究中,我们提出了一种深度生成模型,它将基于随机微分方程(SDE)的扩散模型与预训练的自动编码器相结合。分子发生器能够高效生成针对多种阿片受体(包括 mu、kappa 和 delta)的分子。此外,我们还评估生成分子的 ADMET(吸收、分布、代谢、排泄和毒性)特性,以鉴定药物样化合物。我们开发了一种分子优化方法来增强一些先导化合物的药代动力学特性。

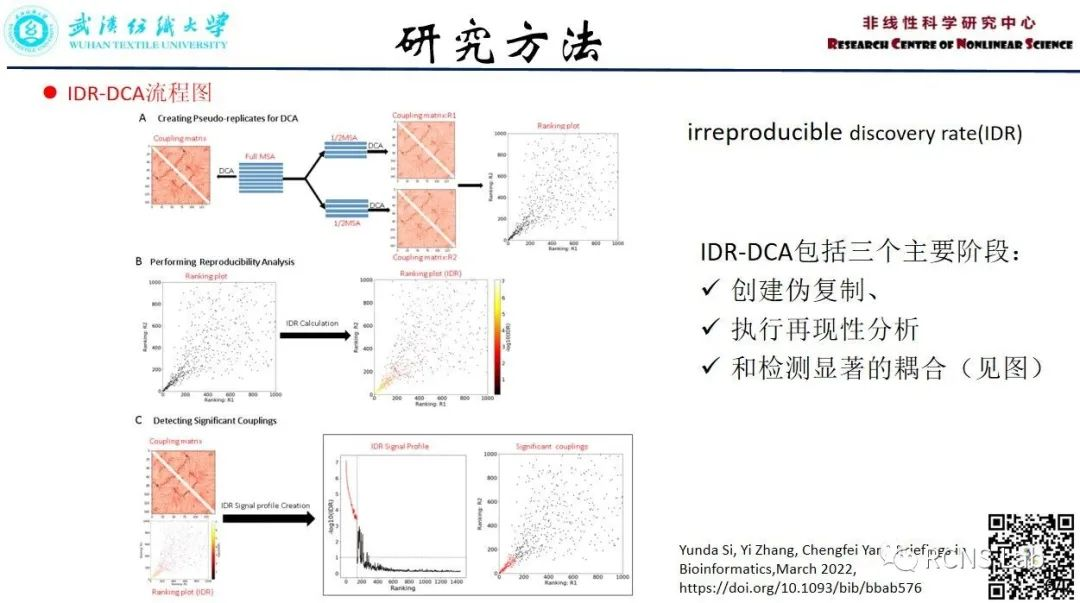
郭成:本次汇报了一篇文献《A Reproducibility Analysis-based Statistical Framework for Residue-Residue Evolutionary Coupling Detection》,直接耦合分析(DCA)已被广泛应用于从同源序列的多序列比对(MSA)中推断残基-残基接触。然而,根据DCA的结果有效地选择残差对进行接触预测是一项重要的任务。在本研究中,我们开发了一个用于显著进化耦合检测的通用统计框架,称为IDR-DCA,它是基于从DCA对手动创建的MSA重复获得的耦合分数的重复性分析。将IDR-DCA应用于选择残基对,进行单体蛋白、蛋白-蛋白相互作用和单体RNA的接触预测,其中应用了3种不同版本的DCA方法。我们证明,在IDR-DCA的应用下,使用通用阈值选择的残基对总是产生稳定的接触预测性能。与仔细应用调整耦合分数截止相比,IDR-DCA总是表现出更好的性能。IDR-DCA的稳健性也通过MSA下采样分析得到了支持。我们进一步证明了应用从IDR-DCA选择的残基对中获得的约束条件来辅助RNA二级结构预测的有效性。
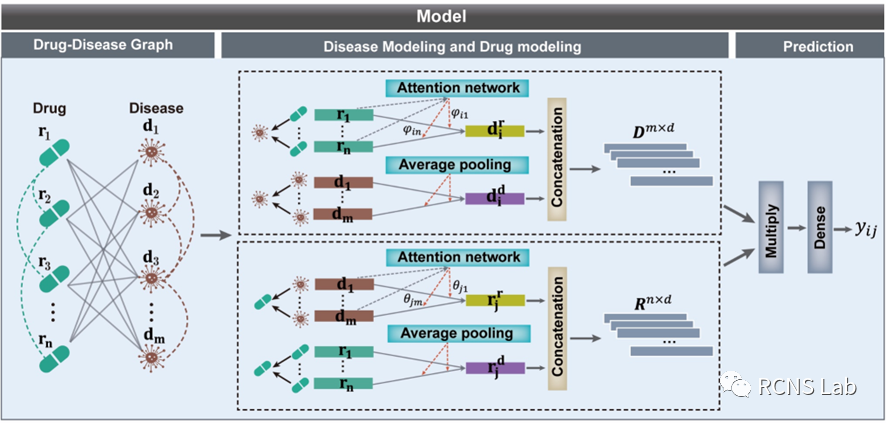
王毅:本次汇报了自己的工作总结,药物重定位是将现有药物重新定向到新的治疗目的的策略,对于加速药物发现至关重要。虽然许多研究都致力于对复杂的药物与疾病关联进行建模,但它们常常忽略了不同节点嵌入之间的相关性。因此,我们提出了一种新颖的加权局部信息增强图神经网络模型,称为DRAGNN,用于药物重新定位。具体来说,DRAGNN首先引入图注意力机制,将注意力系数动态分配给药物和疾病异构节点,增强目标节点信息收集的有效性。为了防止在有限的向量空间中过度嵌入信息,我们省略了自节点信息聚合,从而强调有价值的异构和同质信息。此外,在邻居信息聚合中引入平均池化以增强本地信息,同时保持简单性。然后采用多层感知器来生成最终的关联预测。该模型对药物重新定位的有效性得到了对三个基准数据集的 10 倍 10 倍交叉验证的支持。通过利用多个权威数据源对预测的关联进行分析、分子对接实验以及药物-疾病网络分析,进一步验证,为未来的药物发现奠定坚实的基础。
— 员工汇报照片展示 —



